Cómo se introdujo el virus SARS-CoV-2 en la Provincia de Córdoba
El estudio, firmado por el Consorcio Proyecto PAIS, además analiza la circulación del virus en la provincia. Un trabajo profundo que viene a complementar los presentados anteriormente.
El Consorcio interinstitucional para la Secuenciación del genoma y estudios genómicos de SARS-CoV-2 (Proyecto PAIS), creado desde el Ministerio de Ciencia, Tecnología e Innovación, presentó un reporte donde realiza la caracterización genómica de 93 muestras de individuos con la COVID-19 en la provincia de Córdoba correspondiente al período entre abril de 2020 y enero de 2021. El informe analiza la circulación de SARS-CoV-2 en la provincia, la introducción, circulación y establecimiento de sus linajes y los brotes relevantes.
Mediante análisis filogenético se determinó que todas las secuencias de la provincia de Córdoba pertenecieron al linaje B.1 o sus linajes derivados, que posee amplia distribución tanto a nivel mundial como en Argentina. Las secuencias obtenidas de las 93 muestras fueron asignadas a seis linajes: B.1.499 (45 secuencias: 48,4 %), B.1.1.33 (4 secuencias: 4.3 %) y de su linaje derivado N.3 (37 secuencias: 39.8 %), B.1 (3 secuencias: 3,2 %), B.1.1.222 (3 secuencias: 3,2 %) y B.1.2 (1 secuencia: 1,1 %) (Ver figura 1). Se observa un cambio en la proporción de los linajes en circulación reciente respecto a los previamente detectados en esta región (Ver figura2).
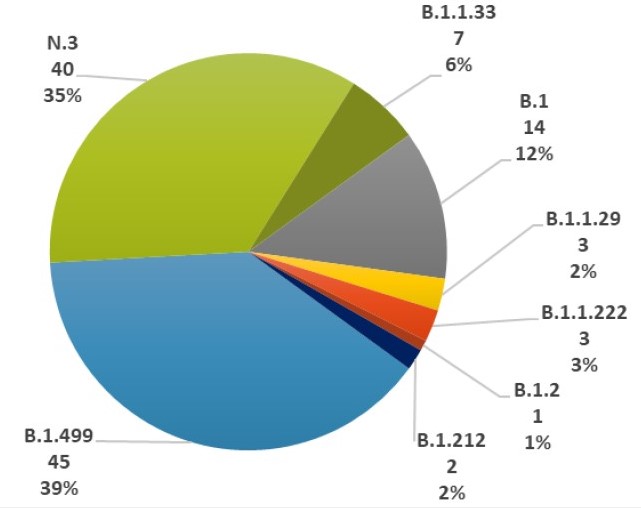
Figura 1. Distribución de Linajes de SARS-CoV-2 hallados en la Provincia de Córdoba (período abril 2020 a enero 2021).
Distribución de linajes encontrados
Durante los meses de marzo-abril predomina el linaje B.1, y se relaciona con introducciones a partir de personas con antecedentes de viaje y a los dos primeros brotes en Córdoba (en Saldán y en un hospital de la ciudad de Córdoba). En los meses de junio-julio se empiezan a detectar los linajes N.3 y B.1.499 en alta frecuencia. Esto sugiere que ambos linajes se establecieron en la provincia de Córdoba a partir de múltiples ingresos posiblemente desde distintas locaciones y que han permanecido circulando en la comunidad. En los meses de noviembre-enero se detectaron por primera vez los linajes B.1.2 y B.1.1.222, así como se detectó nuevamente el linaje B.1.1.33.
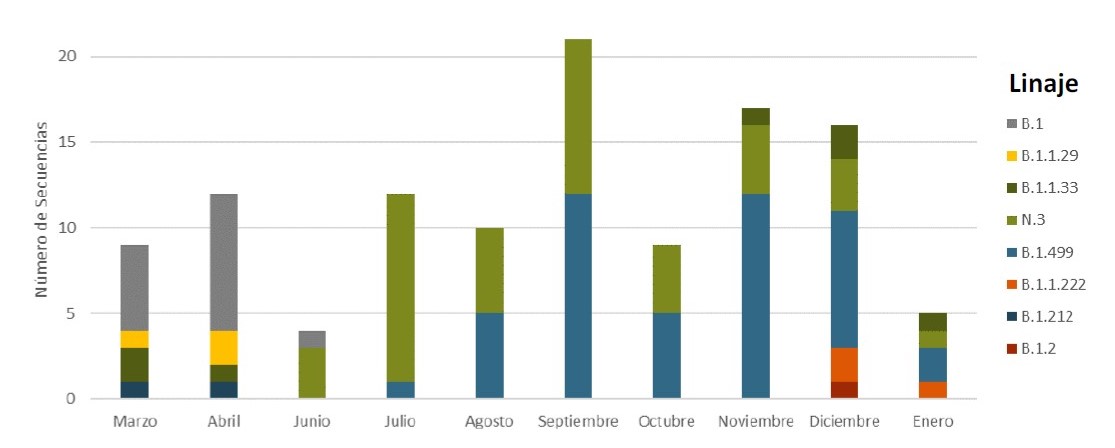
Figura 2. Distribución de linajes de SARS-CoV-2 por mes en el período abril 2020 a enero 2021 en la provincia de Córdoba.
Análisis filogenético de linajes individuales
Linaje B.1: Las primeras secuencias corresponden al primer brote de la COVID-19 en Córdoba ocurrido en el mes de abril de 2020, relacionado a una institución de cuidados de adultos mayores de la Ciudad de Saldán, departamento Colón. Una secuencia relacionada a estas fue obtenida de una muestra proveniente de un brote ocurrido en el barrio El Libertador en el mes de junio. Así, desde una única y temprana introducción en la provincia en abril de 2020, el virus de este grupo ha permanecido en circulación comunitaria en la ciudad de Córdoba hasta al menos el mes de junio de 2020.
Linaje B.1.2: Ha sido reportado en 51 países, pero el 97% de las secuencias corresponden a los Estados Unidos, donde se lo detectó por primera vez en enero de 2020. Una sola secuencia de la provincia de Córdoba perteneció a este linaje, proveniente de un individuo de la ciudad de Córdoba sin antecedente de viaje diagnosticado en diciembre. Esta secuencia se relacionó a un grupo de secuencias de América del Norte (Canadá y Estados Unidos), también correspondientes al mes de diciembre de 2020.
Linaje B.1.1.33: Ha sido descripto como un linaje con origen en Brasil, detectado por primera vez en marzo de 2020 en ese país. Si bien hasta el momento ha sido reportado en 27 países, el 73% de las secuencias corresponden a Brasil y otra gran proporción a países sudamericanos, incluyendo Argentina, Chile y Uruguay.
Las secuencias de este reporte no se relacionaron con las secuencias B.1.1.33 reportadas previamente en Córdoba, por lo que se tratarían de distintas introducciones a la provincia. En particular, las cuatro secuencias de este reporte fueron obtenidas de la ciudad de San Francisco, entre los meses de noviembre y enero, y se entremezclaron con secuencias contemporáneas provenientes de la ciudad de Santa Fe, sugiriendo una cadena de transmisión común del virus en esa región.
Linaje N.3: Del linaje B.1.1.33 surge, al establecerse en la Argentina, el linaje derivado N.3 (alias B.1.1.33.3) detectado por primera vez en abril de 2020. Es reconocido como un linaje argentino, ya que casi la totalidad de las secuencias reportadas en base de datos provienen de nuestro país, con excepción de un reporte de Chile y uno de Hong-Kong.
Las secuencias de Córdoba del linaje N.3 se distribuyeron en dos grandes grupos monofiléticos, que se encuentran cercanos a secuencias de CABA y provincia de Buenos Aires:
-El grupo I incluye 27 muestras de Córdoba obtenidas entre julio y enero; además, este grupo incluyó una secuencia de Merlo, provincia de Buenos Aires. Diecisiete de las 27 secuencias corresponden a la Ciudad de Córdoba, entre las cuales se entremezclan secuencias de otras localidades sin mostrar un patrón geográfico definido. La amplitud temporal y geográfica de las muestras sugiere el establecimiento y circulación comunitaria de este linaje en la provincia.
-El grupo II está conformado únicamente por secuencias de la provincia de Córdoba. Tres secuencias (obtenidas en el mes de julio e informadas previamente) correspondieron a un brote detectado en la Ciudad de Villa Dolores que comenzó en junio. Las nuevas secuencias provienen de muestras tomadas en los meses de junio-julio de 2020 que fueron seleccionadas por su relación con dicho brote. Dos fueron obtenidas de personas que asistieron a un mismo establecimiento bancario al inicio del brote: una perteneciente a un individuo proveniente de CABA que arribó a Villa Dolores con fines laborales (16 de junio de 2020), quien habría arribado a la localidad cordobesa infectado (posible caso índice); la otra muestra proviene de un individuo que viajó La Falda-Villa Dolores-La Falda (18 de junio de 2020). Las dos muestras restantes, correspondieron a distintos momentos del brote (30 de junio y 11 de julio de 2020). Además, este grupo monofilético incluyó secuencias provenientes de localidades ubicadas en el corredor de la ruta 9 (dos de Oncativo, una de Oliva, dos de Villa María y una de la Ciudad de Córdoba), todas obtenidas en el mes de julio.
Los resultados mostraron que todas las secuencias agruparon junto a las secuencias de Córdoba obtenidas previamente formando un cluster monofilético derivado de los virus detectados en el AMBA. Esto confirmaría la introducción de este virus a Villa Dolores desde el AMBA y su posterior diseminación a otras localidades, como La Falda.
Linaje B.1.1.222: Este linaje circula principalmente en Norteamérica (Estados Unidos y México). En Sudamérica ha sido reportado en Brasil y Chile, en casos aislados. Este es el primer reporte del linaje en Argentina.
Las tres secuencias de este linaje provienen de la ciudad de Córdoba de los meses de diciembre 2020 y enero 2021, dos de ellas provenían de individuos que concurrieron a un mismo evento social. Todas comparten un ancestro común cercano y el grupo presenta alto soporte estadístico, lo que sugiere que se trataría de una única introducción a la provincia. La secuencia filogenéticamente más cercana corresponde al Reino Unido, agrupando también con secuencias de Estados Unidos y México.
Linaje B.1.499: Este linaje, plenamente argentino y detectado inicialmente en marzo del 2020, ha sido reportado en diferentes provincias del país, principalmente en la región del AMBA. El mismo fue el mayoritario en las secuencias analizadas en este reporte (48,4%), se detectó en varias regiones de la provincia y en diferentes momentos de la epidemia, sugiriendo su establecimiento y circulación comunitaria en la provincia de Córdoba. Llamativamente, fue el único linaje encontrado en la localidad de Río Cuarto. Las secuencias de Córdoba se distribuyeron en al menos cinco grupos distintos y dos secuencias aisladas, lo que sugiere múltiples introducciones a la provincia, posiblemente desde distintas regiones del país:
-El grupo I está conformado por cuatro secuencias de Córdoba y una de la CABA.
-El grupo II incluye dos secuencias que comparten un ancestro en común con secuencias de la provincia de Santa Fe.
-El grupo III incluye tres secuencias de Córdoba, relacionadas con secuencias de Neuquén y Río Negro, el sur de la provincia de Buenos Aires (Patagones) y una de la CABA.
-El grupo IV está conformado enteramente por secuencias de Córdoba obtenidas entre agosto 2020 y enero 2021 e incluye principalmente secuencias de ciudad de Córdoba (n=6), Unquillo (n=2), Villa Dolores (n=1) y San Francisco (n=1).
-El grupo V es el más numeroso e incluyó a 21 secuencias de Córdoba, dos grupos internos de secuencias de La Pampa y una secuencia de Santa Fe.
Finalmente, se observaron dos secuencias aisladas. Una de ellas proviene de Alta Gracia y se incluyó en un grupo de secuencias del NOA, mientras que la otra corresponde a la ciudad de Marcos Juárez y agrupó con secuencias de la ciudad de Rosario.
Conclusiones
Todas las secuencias corresponden al linaje B.1 o sus derivados, lo que es compatible con la amplia distribución que presenta a nivel mundial y es congruente con lo observado anteriormente para Argentina y para la mayoría de los países de América del Sur.
El análisis comparativo con la primera etapa de estudio de secuencias de esta región del país permitió establecer que se registró un cambio en los linajes en circulación. El linaje B.1.499 fue el mayoritario, mientras que el linaje B.1.1.33, detectado en los casos inicialmente estudiados asociados a viajeros provenientes de Brasil, y su derivado N.3, continuaron circulando en alta frecuencia. Esto sugiere que ambos linajes se establecieron en la provincia de Córdoba a partir de múltiples ingresos posiblemente desde distintas locaciones y algunos de ellos han permanecido circulando en la comunidad. El linaje B.1, responsable de los dos primeros brotes en Córdoba solo fue detectado hasta junio 2020, lo que indicaría que su circulación podría haber sido desplazada por otros linajes.
Participaron del reporte por el Laboratorio Central, Ministerio de Salud de la provincia de Córdoba, Gabriela Barbás, Gonzalo Castro, Paola Sicilia y Laura López; por el Instituto de Virología “Dr. J. M. Vanella” Facultad de Ciencias Médicas-Universidad Nacional de Córdoba, Viviana Ré, María Belén Pisano; del Nodo de secuenciación de Córdoba, Franco Fernández, Nathalie Marquez y Humberto Debat (IPAVE-INTA-CIAP); del Nodo de secuenciación de Rafaela-Santa Fe, Matías Irazoqui, Ariel Amadío (EEA Rafaela-INTA-CONICET) y del Nodo evolución, Carolina Torres, Débora Marcone, Laura Mojsiejczuk, María Dolores Blanco Fernández, Andrés Culasso, Humberto Debat, Mariana Viegas.
Proyecto PAIS
Creado desde el Ministerio de Ciencia, Tecnología e Innovación de la Nación, el Consorcio interinstitucional para la Secuenciación del genoma y estudios genómicos de SARS-CoV-2 (Proyecto PAIS) está conformado por grupos de investigación de diferentes instituciones con el objetivo de secuenciar el genoma y realizar demás estudios genómicos del SARS-CoV-2.
Coordinado por la Dra. Mariana Viegas del Laboratorio de Virología Hospital de Niños Ricardo Gutiérrez, el equipo trabaja articulada y colaborativamente para realizar estudios genómicos de SARS-CoV-2 en nuestro país y aportar tanto al conocimiento local como a la base de datos global de circulación viral GISAID (Global Initiative on Sharing All Influenza Data). Entre sus objetivos se encuentran la secuenciación de los genomas circulantes de SARS-CoV-2 en distintas regiones de nuestro país, el análisis a gran escala de secuencias, ensamblado de genomas, análisis filogenéticos y filogeográficos, epidemiología y evolución molecular.